文章目录
写在前面
本人初步接触深度学习与医学图像分割领域,第一个项目就是运用nnUNet网络对BrainTumour的数据集进行分割,在学习的过程中,学了很多资料,踩了很多坑,也解决了很多bug,在此把自己的学习经验分享出来,希望有需要的同学可以少走一些弯路。
本篇博客的操作均在服务器的linux系统(Ubuntu)上进行。作者的建议也是使用linux系统。
本篇文章使用的数据集为Medical Segmentation Decathlon比赛的Task01_BrainTumour数据集,如果你想训练自己的数据集,请参考我的另一篇博客。
才疏学浅,如有错误,欢迎批评指正!!
nnUNet是什么?
nnU-Net是由德国癌症研究中心、海德堡大学以及海德堡大学医院研究人员(Fabian Isensee, Jens Petersen, Andre Klein)提出来的一个自适应任何新数据集的医学影像分割框架,该框架能根据给定数据集的属性自动调整所有超参数,整个过程无需人工干预。仅仅依赖于朴素的U-Net结构(就是原始U-Net)和鲁棒的训练方案,nnU-Net在六个得到公认的分割挑战中实现了最先进的性能。
- 关于nnUNet,如果你想更加深入的了解,我推荐看一下这篇博客论文解读- nnU-Net: Self-adapting Framework for U-Net-Based Medical Image Segmentation(附实现教程)_Tina姐的博客-CSDN博客,是比较概括性的讲解,更深入的学习还是建议精读论文。
一、配置虚拟环境
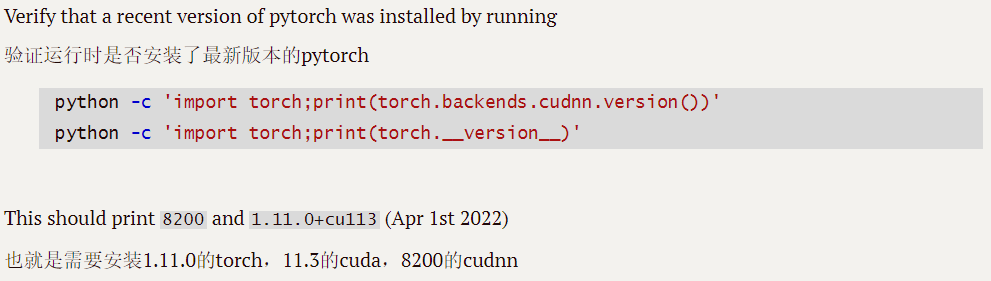
首先,我们想实现nnUNet的使用,需要配置类似如下的环境:
-
上图所示为nnUNet的官方readme文档建议的环境配置,如果你对cuda,cudnn,torch之类的词语还不算熟悉,可以看一下我的另一篇博客用人话讲解深度学习中CUDA,cudatookit,cudnn和pytorch的关系_江江ahh的博客-CSDN博客
-
至于为什么要在虚拟环境中安装nnUNet,当我们在安装包的时候,
pip install django安装一个包会附带安装数个其他关联的包。但是当我们删除这个包的时候,我们之后仅仅删除这一个包,安装时附带的其他包并不会删除。**简单来说,当我们的环境使用时间长的时候,我们的包是很难进行管理的。**所以非常建议在一个全新的虚拟环境中来完成接下来的操作!!! -
如果你对虚拟环境还比较陌生,建议看一下这位大佬的视频安装不算完事,只有理解了虚拟环境才算真正掌握 Python 环境_哔哩哔哩_bilibili
-
当你了解虚拟环境的作用以后,可以参考我的另一篇博客来配置nnUNet所需的环境在conda虚拟环境中配置cuda+cudnn+pytorch深度学习环境(新手必看!简单可行!)_江江ahh的博客-CSDN博客_在虚拟环境中安装cuda
二、安装nnUNet框架
此时,你应该已经配置好了自己的虚拟环境并且可以打印出类似上图的环境版本(可以更高,但最好不要太低),注意,后续的操作均要在你激活你想要使用的那个虚拟环境的前提下进行!!!!
1.安装nnUNet
根据readme文档,这里应该有两种方案可供选择:
(1)用作标准化基线、开箱即用的分割算法或使用预训练模型进行推理:
pip install nnunet(2)用作集成框架(这将在您的计算机上创建nnU-Net代码的副本,以便您可以根据需要对其进行修改)
你想把nnUNet的文件夹放在哪,就在哪个路径下运行这些命令!
git clone https://github.com/MIC-DKFZ/nnUNet.gitcd nnUNetpip install -e .#最后这个点也不能忽略对于我来说,因为我后续是要改网络代码的,所以我选择第二种方法,下面详细说明一下这三行命令都是什么意思:
git clone其实就是把人家github上的代码克隆过来,这一步其实和直接复制粘贴代码文件是一样的,总共也就1Mb
cd nnUNet不用说了,就是进入文件夹
其实这个时候就已经有一个nnUNet完整的文件夹了,里面包含这些东西:
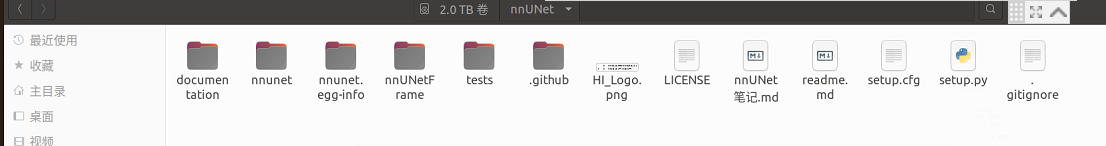
最后pip install -e .相当于python setup.py,也就是运行上图这个setup.py文件
这个文件是用来干什么的呢?
- 安装nnUNet需要的python包
- 向终端添加几个新命令。这些命令用于运行整个nnU-Net pipeline。您可以从系统上的任何位置执行它们。所有nnU-Net命令都带有前缀“nnUNet_”,以便于识别。
这一步我遇到的两个问题:
(1)从github上git clone代码的时候速度慢的离谱(20kb/s),这个问题主要是github的域名在国内被限制了,网上有很多方法解决,比如下面这个网址可以参考https://www.jianshu.com/p/d58ab49ba98b/
(2)运行pip命令的时候速度很慢,这个问题导致我运行pip install -e .的时候超时报错了,同样给出解决方法的链接解决Linux,Ubuntu下使用python包管理工具pip命令安装和下载包速度很慢、失败或者connection timeout等问题_一点儿也不萌的萌萌的博客-CSDN博客_linux 下载python failed: connection timed out.
2.安装隐藏层hiddenlayer(可选)
隐藏层使nnU-net能够给出其生成的网络拓扑图(后面会细说),安装命令如下(这是一整行命令,请务必一起复制粘贴)
pip install --upgrade git+https://github.com/FabianIsensee/hiddenlayer.git@more_plotted_details#egg=hiddenlayer这里我遇到的唯一问题就是上面说的pip命令速度太慢,也是根据上述解决方案来解决的。
三、数据集准备
nnUNet对于你要训练的数据是有严格要求的,这第一点就体现在我们保存数据的路径上,请初学者务必按照我下面的样式来创建相应的文件夹并存入数据!!!
第一步:你现在应该有一个名为nnUNet的文件夹(上面有图),进入它,在里面创建一个名为nnUNetFrame的文件夹
第二步:在nnUNetFrame文件夹中创建一个名为DATASET的文件夹,后面我们会用它来存放数据
第三步:在DATASET文件夹中创建三个文件夹,它们分别是nnUNet_raw,nnUNet_preprocessed,nnUNet_trained_models
第四步:进入上面第二个文件夹nnUNet_raw,创建nnUNet_cropped_data文件夹和nnUNet_raw_data文件夹,右边存放原始数据,左边存放crop以后的数据。
第五步:进入右边文件夹nnUNet_raw_data,创建一个名为Task01_BrainTumour的文件夹(解释:这个Task01_BrainTumour是nnUNet的作者参加的一个十项全能竞赛的子任务名,也是我要实践的分割任务,类似的还有Task02_Heart,就是分割心脏的。如果你想分割自己的数据集,建议Task_id从500开始,这样以确保不会与nnUNet的预训练模型发生冲突(ID不能超过999))
第六步:将下载好的公开数据集或者自己的数据集放在上面创建好的任务文件夹下,下面还以Task01_BrainTumour竞赛为例,解释下数据应该怎么存放和编辑:
- 进入这个网站http://medicaldecathlon.com/.下载对应的数据集(<–网上学科议建<–),取代上面你自己创建的Task01_BrainTumour文件夹。
- 你会发现目录是这个样子的:json文件是对三个文件夹内容的字典呈现(关乎你的训练),imagesTr是你的训练数据集,打开后你会发现很多的有序的nii.gz的训练文件,而labelsTr里时对应这个imagesTr的标签文件,同样为nii.gz。目前只能是nii.gz文件,nii文件都不行。训练阶段的imageTs文件夹先不管,其实这个文件夹出现在任何位置都可以。(解释:nnUNet使用的是五折交叉验证,并没有验证集)
四、设置nnUNet读取文件的路径
nnUNet是如何知道你的文件存放在哪儿呢,当然要在环境中创建一个路径,这个路径你唯一需要更改的是/nnUNet之前的路径,因为后面的路径你和我是一样的。
第一步:在home目录下按ctrl + h,显示隐藏文件
第二步:找到.bashrc文件,打开
第三步:在文档末尾添加下面三行,保存文件。
export nnUNet_raw_data_base="/home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw"export nnUNet_preprocessed="/home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_preprocessed"export RESULTS_FOLDER="/home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_trained_models" 第四步:在home下打开终端,输入source .bashrc来更新该文档
现在nnUNet已经知道怎么读取你的文件了。
五、数据集转换
1.数据集转换是什么,为什么要进行数据集转换?
nnUNet要求将原始数据转换成特定的格式,以便了解如何读取和解释数据。
每个分割数据集存储为单独的“任务”。命名包括任务与任务ID,即三位整数和相关联的任务名称。
比如Task001_BrainTumour的任务名称为“脑瘤”,任务ID为1。
在每个任务文件夹中,预期的结构如下:
Task001_BrainTumour/├── dataset.json├── imagesTr├── (imagesTs)└── labelsTr图像可能具有多种模态,这对于医学图像来说尤其常见。
nnU-Net通过其后缀(文件名末尾的四位整数)识别成像模态。因此,图像文件必须遵循以下命名约定:case_identifier_XXXX.nii.gz。
这里,XXXX是模态标识符。dataset.json文件中指定了这些标识符所属的模态。
标签文件保存为case_identifier.nii.gz
此命名方案产生以下文件夹结构。用户有责任将其数据转换为这种格式!
下面是MSD的第一个任务的示例:BrainTumor。每个图像有四种模态:FLAIR(0000)、T1w(0001)、T1gd(0002)和T2w(0003)。请注意,imagesTs文件夹是可选的,不必存在。
nnUNet_raw_data_base/nnUNet_raw_data/Task001_BrainTumour/├── dataset.json├── imagesTr│ ├── BRATS_001_0000.nii.gz│ ├── BRATS_001_0001.nii.gz│ ├── BRATS_001_0002.nii.gz│ ├── BRATS_001_0003.nii.gz│ ├── BRATS_002_0000.nii.gz│ ├── BRATS_002_0001.nii.gz│ ├── BRATS_002_0002.nii.gz│ ├── BRATS_002_0003.nii.gz│ ├── BRATS_003_0000.nii.gz│ ├── BRATS_003_0001.nii.gz│ ├── BRATS_003_0002.nii.gz│ ├── BRATS_003_0003.nii.gz│ ├── BRATS_004_0000.nii.gz│ ├── BRATS_004_0001.nii.gz│ ├── BRATS_004_0002.nii.gz│ ├── BRATS_004_0003.nii.gz│ ├── ...├── imagesTs│ ├── BRATS_485_0000.nii.gz│ ├── BRATS_485_0001.nii.gz│ ├── BRATS_485_0002.nii.gz│ ├── BRATS_485_0003.nii.gz│ ├── BRATS_486_0000.nii.gz│ ├── BRATS_486_0001.nii.gz│ ├── BRATS_486_0002.nii.gz│ ├── BRATS_486_0003.nii.gz│ ├── BRATS_487_0000.nii.gz│ ├── BRATS_487_0001.nii.gz│ ├── BRATS_487_0002.nii.gz│ ├── BRATS_487_0003.nii.gz│ ├── BRATS_488_0000.nii.gz│ ├── BRATS_488_0001.nii.gz│ ├── BRATS_488_0002.nii.gz│ ├── BRATS_488_0003.nii.gz│ ├── BRATS_489_0000.nii.gz│ ├── BRATS_489_0001.nii.gz│ ├── BRATS_489_0002.nii.gz│ ├── BRATS_489_0003.nii.gz│ ├── ...└── labelsTr ├── BRATS_001.nii.gz ├── BRATS_002.nii.gz ├── BRATS_003.nii.gz ├── BRATS_004.nii.gz ├── ...如果对于数据集转换这件事还是不明白,打开你的nnUNet文件夹,在/nnUNet/documentation/文件夹下找到dataset_conversion这个文件来进一步学习。
2.运行数据集转换的命令
依旧以Task01_BrainTumour为例:
nnUNet_convert_decathlon_task -i /home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task01_BrainTumour转换操作完成以后,你会发现在你的Task01_BrainTumour文件夹旁边,出现了一个Task001_BrainTumour文件夹,打开看一下,里面的格式应该和我上面展示的一样。
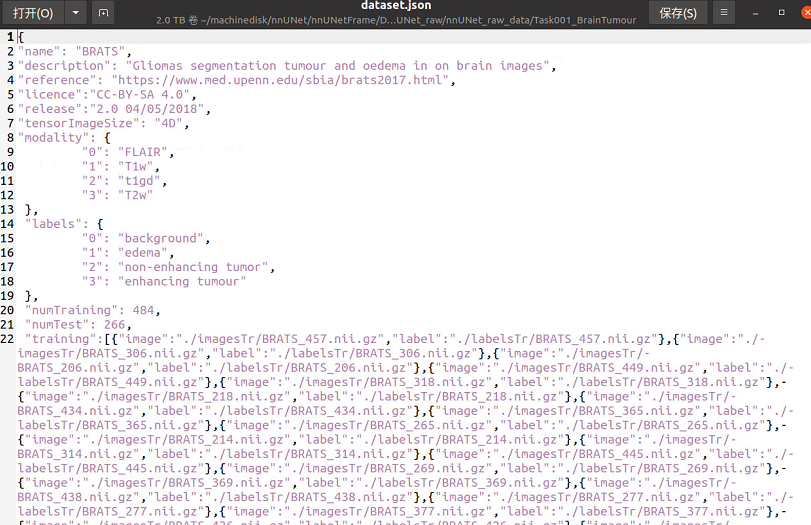
3.关于dataset.json文件
这个文件包含你的训练数据信息和任务信息,如果你按照我的建议下载了Task01的数据集,那里面是包含dataset.json文件的,如果你有训练自己的数据集的需求,在我的另一篇博客里会有详细的说明。
六、数据预处理
nnUNet_plan_and_preprocess -t 1 --verify_dataset_integrity只需要一行命令,因为我们的Task_id是1,所以这里的数字就是1。这个过程会消耗很多的时间,速度慢的原因在于对要进行插值等各种操作。
根据nnUNet框架,三维医学图像分割的通用预处理可以分为四步,分别是数据格式的转换,裁剪crop,重采样resample以及标准化normalization。如果你想进一步学习,推荐学习这篇文章如何针对三维医学图像分割任务进行通用数据预处理:nnUNet中预处理流程总结及代码分析 - 知乎 (zhihu.com)
运行“nnUNet_plan_and_preprocess”将使用预处理数据填充文件夹。
我们将在nnUNet_preprocessed/Task001_BrainTumour中找到这条命令的输出结果。使用2D U-Net以及所有适用的3D U-Net的预处理数据创建子文件夹。它还将为2D和3D配置创建“plans”文件(结尾为.pkl)。这些文件包含生成的分割 pipeline 配置,将由nnUNetTrainer读取(见下文)。请注意,预处理的数据文件夹仅包含训练案例。测试图像没有经过预处理。测试集的预处理将会在推理过程中实时进行。
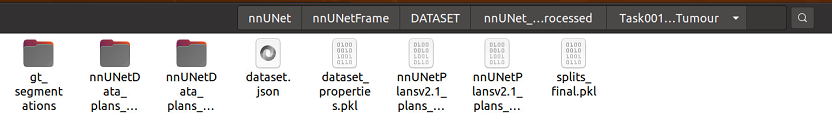
另外,`–verify_dataset_integrity”应至少在给定数据集上首次运行命令时运行。这将对数据集执行一些检查,以确保其与nnU-Net兼容。如果此检查通过一次,则可以在以后的运行中省略。如果您遵守数据集转换指南(请参见上文),那么这条命令一定会通过的。
七、模型训练
1.写在训练前:更改epoch
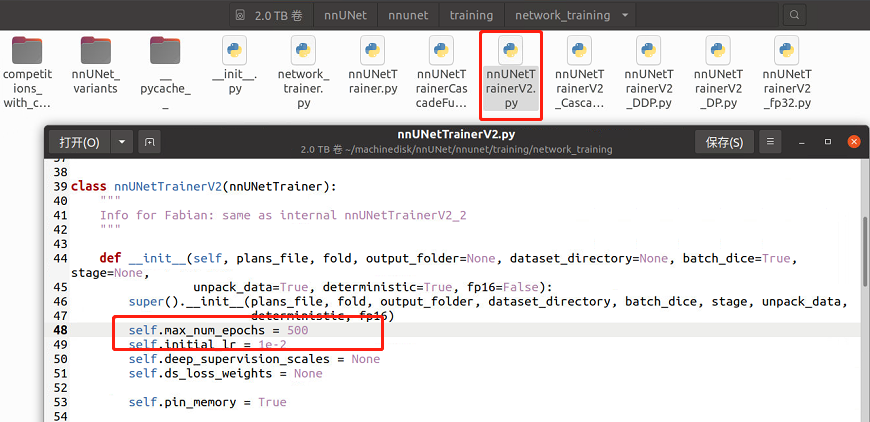
nnUNet默认原始的epoch是1000,这太久了,我们打开nnUNet/nnunet/training/network_training/nnUNetTrainerV2.py
第48行的max_epoch来修改epoch
2.关于训练的运行命令
nnU-Net在5倍交叉验证中训练所有U-Net配置。这使nnU-Net能够确定训练数据集的后处理和集合(参见下文)。
如果你不清楚什么是K折交叉验证,可以学习这篇博客【技术分享】什么是K折交叉验证?_AISec盐究员的博客-CSDN博客_k折交叉验证
我们在数据预处理那一步的时候创建了三个U-Net配置:2D U-Net、对全分辨率图像进行操作的3D U-Net以及3D U-Net级联,其中级联的第一个U-Net在下采样图像中创建粗分割图,然后由第二个U-Net进行细化。我们在训练的时候可以自由选用它们。
训练模型使用“nnUNet_train”命令完成。命令的一般结构为:
nnUNet_train CONFIGURATION TRAINER_CLASS_NAME TASK_NAME_OR_ID FOLD --npz (additional options)- CONFIGURATION是一个字符串,用于标识所请求的U-Net配置。
- TRAINER_CLASS_NAME是model trainer的名称。如果您实施定制trainers(nnU-Net作为一个框架),您可以在此处指定您的定制trainers。
- TASK_NAME_OR_ID指定应训练的数据集,FOLD指定训练的是5倍交叉验证的哪一倍。
- “–npz”使模型在最终验证期间保存softmax输出。它仅适用于计划在之后运行“nnUNet_find_best_configuration”的训练
(这是nnU Nets自动选择最佳性能(集合)配置,见下文)。
对于我们的Task01来说,应该运行的命令如下
nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz- 3d_fullres代表我们选用对全分辨率图像进行操作的3D U-Net
- nnUNetTrainerV2是我们选用的训练器
- 1代表你的任务ID
- 0代表五折交叉验证中的第0折
下面给出各种配置的nnUNet网络需要的训练命令
2D U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 2d nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz3D full resolution U-Net 3D全分辨率U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_fullres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz3D U-Net cascade 3D U-net级联
①3D low resolution U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_lowres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz②3D full resolution U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_cascade_fullres nnUNetTrainerV2CascadeFullRes TaskXXX_MYTASK FOLD --npz注意,级联的3D全分辨率U-Net需要预先完成低分辨率U-Net的five folds!
3.训练结果
训练后的模型将写入RESULTS_FOLDER/nnUNet文件夹。对于我们的项目来说,就是会存在/home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_trained_models/nnUNet这个路径下。
每次训练都会获得一个自动生成的输出文件夹名称,根据我们的训练配置,我们会得到3d_fullres/Task001_BrainTumour这个文件夹。关于它的树状图如下(为简洁起见,有些文件仅在一个文件夹下详细展开):
RESULTS_FOLDER/nnUNet/├── 2d│ └── Task001_BrainTumour│ └── nnUNetTrainerV2__nnUNetPlansv2.1│ ├── fold_0│ ├── fold_1│ ├── fold_2│ ├── fold_3│ └── fold_4├── 3d_cascade_fullres├── 3d_fullres│ └── Task001_BrainTumour│ └── nnUNetTrainerV2__nnUNetPlansv2.1│ ├── fold_0│ │ ├── debug.json│ │ ├── model_best.model│ │ ├── model_best.model.pkl│ │ ├── model_final_checkpoint.model│ │ ├── model_final_checkpoint.model.pkl│ │ ├── network_architecture.pdf│ │ ├── progress.png│ │ └── validation_raw│ │ ├── BRATS_010.nii.gz│ │ ├── BRATS_010.pkl│ │ ├── BRATS_018.nii.gz│ │ ├── BRATS_018.pkl│ │ ├── summary.json│ │ └── validation_args.json│ ├── fold_1│ ├── fold_2│ ├── fold_3│ └── fold_4└── 3d_lowres如果你的训练成功了,应该会得到和我下图一样的结果
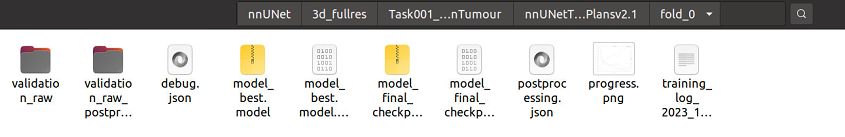
下面详细讲讲这些训练后得到的文件都是什么
-
debug.json:包含用于训练此模型的蓝图和推断参数的摘要。不容易阅读,但对调试非常有用。
-
model_best.model/model_best.model.pkl:训练期间识别的最佳模型的检查点文件。
-
model_final_checkpoint.model/model_final_checkpoint.model.pkl:最终模型的检查点文件(训练结束后)。这是用于验证和推理的。
-
networkarchitecture.pdf(仅当安装了hiddenlayer时!):一个pdf文档,其中包含网络架构图。
-
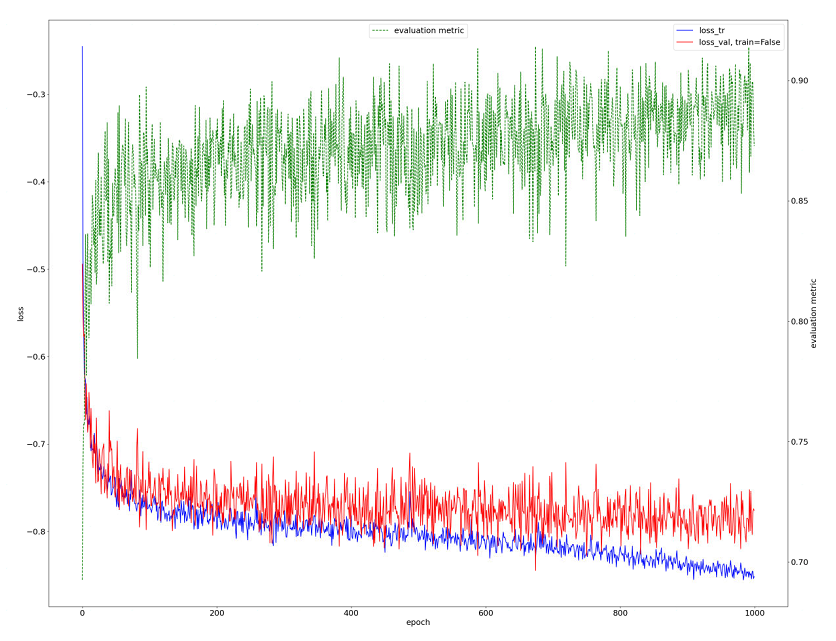
progress.png:训练期间训练(蓝色)和验证(红色)损失的图。还显示了评估指标的近似值(绿色)。这个近似值是前景类的平均Dice分数。
-
validation_raw:在这个文件夹中是训练完成后预测的验证案例。summary.json包含验证度量(文件末尾提供了所有情况的平均值)。
-
training_log:训练过程中不断打印,nnunet的loss函数默认是趋向-1的,也就是说在训练的过程中,我们通过每轮训练的日志可以查看到每轮的loss函数,这个数值应该是负数,而且越趋向于-1,效果越好。
现在我们想看看我们训练的结果怎么样,有两种方法:
第一种是打开progress.png来从图像上直观的感受一下,它大概长这样:
第二种是打开validation_raw/summary.json,从里面我们不仅可以看到对每一个验证数据的评价,更可以在最末尾看到它们的平均值。
这里我们主要关注这个Dice分数,之前说到了脑瘤数据集分为四个模态,而labels的数量也是4(参见dataset.json):
"labels": { "0": "background", "1": "edema", "2": "non-enhancing tumor", "3": "enhancing tumour"包含背景在内的4个标签,分别是背景、坏疽(NET,non-enhancing tumor)、浮肿区域(ED,peritumoral edema)、增强肿瘤区域(ET,enhancing tumor),如下图,它们的平均dice分数约为0.9994,0.8770,0.7780,0.8728

八、确定最佳U-Net配置
本文只说明了3d_fullres的训练,完整的nnUNet流程还需要跑2d和3d级联的,然后进行三种的择优。不过从实际性能来说,一般3d级联≥3d>2d,是否跑其他两种需要自己考虑。
训练完所有模型后,使用以下命令自动确定用于测试集预测的U-Net配置:
nnUNet_find_best_configuration -m 2d 3d_fullres 3d_lowres 3d_cascade_fullres -t 1- 所有指定配置都需要完成所有的5折训练!
- 对于未配置级联的数据集,请改用“-m 2d 3d_fullres”。如果您只想探索配置的某些子集,可以使用“-m”命令指定。
所以说,如果你训练完所有模型,才可以使用这一步,如果没有,那可以直接推理
九、运行推理
1.准备测试集
让我们回到你刚刚做好数据集转换的那个数据集:/home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour,在里面创建inferTs这个文件夹,用于存放待推理测试集的推理结果。然后,我会选择将原本的imagesTs重命名为imagesTs0,它是我们下载数据集时给我们的几十个测试集,然后新建一个imagesTs,里面只放一个测试集。(解释:这个道理其实很明显,推理太多数据集太久了,先用一个试试)
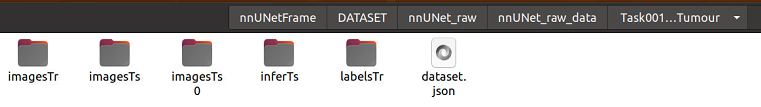
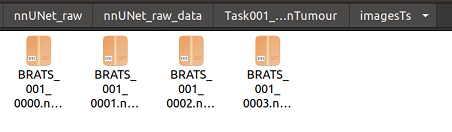
这个时候我们在imagesTs里存放的待推理的测试集,它的格式应该是经过数据集转换那一步的格式,忘记了的话可以翻上去看一下,如下图所示,四个模态都要有,且重命名过:
2.运行推理的最简单方法是简单地使用下面这一条命令:
nnUNet_predict -i 要预测数据的文件夹路径 -o 输出文件夹路径 -t 1 -m 3d_fullres -f 0对于我们这个项目,它就应该是:
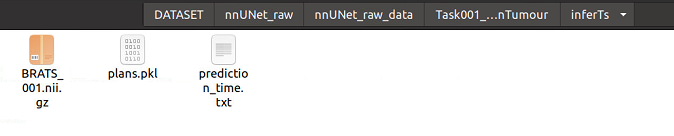
nnUNet_predict -i /home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/imagesTs/ -o /home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/inferTs -t 1 -m 3d_fullres -f 0你会在inferTs里得到模型生成的预测结果
3.如果你想集成多个模型的推理结果
如果你已经训练出了多个模型,并且像上一步一样进行了推理,现在你想集成它们的推理结果,首先需要在上面的命令后面加入“–save_npz”。`–save_npz”将使命令保存softmax概率以及需要大量磁盘空间的预测分割掩码。
然后请为每个配置选择单独的“OUTPUT_FOLDER”!
最后使用以下命令集合来自多个配置的预测:
nnUNet_ensemble -f FOLDER1 FOLDER2 ... -o OUTPUT_FOLDER -pp POSTPROCESSING_FILE您可以指定任意数量的文件夹,但请记住,每个文件夹都需要包含由“nnUNet_predict”生成的npz文件。对于集成,还可以指定一个文件,告诉命令如何进行后处理。
这些文件是在运行“nnUNet_find_best_configuration”时创建的,位于相应的训练模型目录中(RESULTS_FOLDER/nnUNet/CONFIGURATION/TaskXXX_MYTASK/TRAINER_CLASS_NAMEPLANS_FILE_IDENTIFIER/postprocessing.json or
RESULTS_FOLDER/nnUNet/ensembles/TaskXXX_MYTASK/ensemble_XYZ–XY__Z/postprocessing.json).
您也可以选择不提供文件(只需省略-pp),nnU-Net将不会运行后处理。
因此,在运行集成推理之前,必须对所有5个折进行训练。在推理开始时,将打印找到的可用的nnU-Net folds列表。
十、评估推理结果
依旧是一行命令完成
nnUNet_evaluate_folder -ref 金标准文件夹 -pred 预测结果文件夹 -l 1 2 3-l 表示要计算的label的类别,正常就是背景-0肿瘤-1,所以设置1,如果有两类就是 -l 1 2,以此类推,所以我们这里是1 2 3
这个是nnUNet自带的评估命令,计算分割DSC,可以不用这个,另写代码去算需要的评估指标即可
我在相应路径下创建了seg_0文件夹和seg_p文件夹,分别用来存放金标准和预测结果

运行如下命令
nnUNet_evaluate_folder -ref /home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/seg_0 -pred /home/work/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/seg_p -l 1 2 3在seg_p文件夹中,我们会得到和训练结果文件夹里validation_raw/summary.json一样格式的summary.json文件,它包含了每一个评估数据的指标,文档末尾是其平均值。
参考资料
https://blog.csdn.net/u014264373/article/details/116792649
(四:2020.07.28)nnUNet最舒服的训练教程(让我的奶奶也会用nnUNet(上))(21.04.20更新)_花卷汤圆的博客-CSDN博客_nnuet
(五:2020.07.31)nnUNet最简单的推理教程(让我的奶奶也会用nnUNet(下))_花卷汤圆的博客-CSDN博客_nnunet推理
医学图像分割 3D nnUNet全流程快速实现_打南边来了个阿楠的博客-CSDN博客
医学图像分割 3D nnUNet全流程快速实现_打南边来了个阿楠的博客-CSDN博客
码字不易,您的点赞是对我最好的支持,如果有问题也欢迎在评论区或私信与我交流!
来源地址:https://blog.csdn.net/m0_68239345/article/details/128886376




