这篇“怎么使用R语言ComplexHeatmap绘制复杂热图heatmap”文章的知识点大部分人都不太理解,所以小编给大家总结了以下内容,内容详细,步骤清晰,具有一定的借鉴价值,希望大家阅读完这篇文章能有所收获,下面我们一起来看看这篇“怎么使用R语言ComplexHeatmap绘制复杂热图heatmap”文章吧。
一 载入R包 数据
1.1 载入ComplexHeatmap包,数据
为更贴近生信使用场景,直接使用内置的基因表达数据
library(ComplexHeatmap)expr = readRDS(paste0(system.file(package = "ComplexHeatmap"), "/extdata/gene_expression.rds"))#查看数据str(expr)expr[1:4,c(1:4,25:27)]
拿到一个新数据后,除了检查[1:4,1:4]外,也许还需要看看最后几列,另外还需要观察列名称的规律。
去除最后几列,或者只选取列名字包含cell的(TCGA数据处理中也会经常遇到)
mat = as.matrix(expr[, grep("cell", colnames(expr))])1.2 绘制最简单的热图
Heatmap(mat)二 常见“表型”注释
文献中经常见到的就是在热图的top 或者 bottom位置添加样本的变异信息,临床信息等的注释,本节介绍如何实现以及常见的设置。
读入注释文件
anno <- read.csv("anno.csv",header = T) #非真实数据,随便设置head(anno) sample stage age#1 s1_cell01 1 56#2 s2_cell02 2 43#3 s3_cell03 2 63#4 s4_cell01 3 23#5 s5_cell02 1 8#6 s6_cell03 3 32.1 添加注释,且设置颜色
1.1 颜色设置
1) 连续变量:指定色系,根据变量范围设置颜色范围
col_fun2 <- colorRamp2( c(0, 50, 100), #根据值的范围设置 c("#ff7f00", "white", "#1f78b4"))2)分类变量:直接指定颜色编码
#stage = c("1" = "red", "2" = "green", "3" = "blue" , "4" = "orange") #分类1.2 添加注释
使用HeatmapAnnotation函数进行注释,添加待注释的内容
ha <- HeatmapAnnotation( age = anno$age, stage = anno$stage, col = list( age = col_fun2 , #连续 stage = c("1" = "red", "2" = "green", "3" = "blue" , "4" = "orange") #分类 ))1)注释位置
#指定注释位置 ,示例为top_annotation,此外可选 bottom_annotation ,right_annotation ,left_annotation
Heatmap( mat, top_annotation = ha )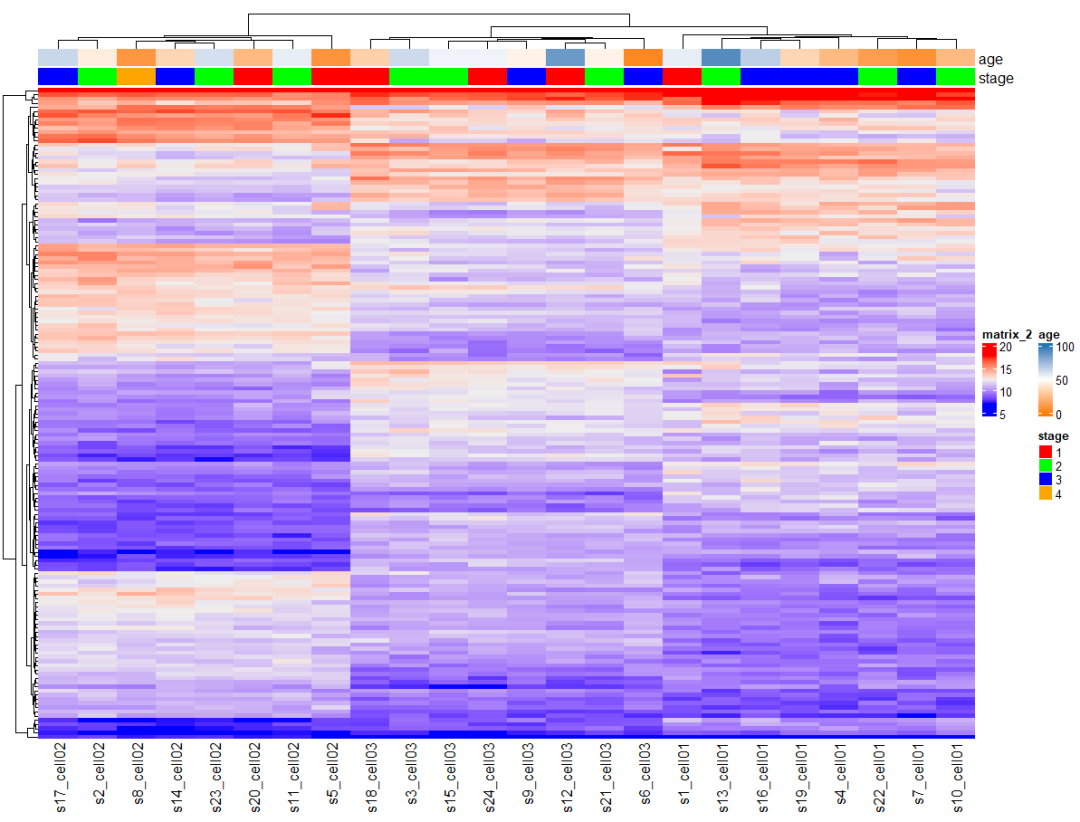
热图上面注释样本的临床等信息,实现!
2) 指定多个注释位置
当需要注释的内容较多时候,可以选择在不同的位置。需要预先根据待注释的位置进行指定
column_ha <- HeatmapAnnotation( bar1 = anno_barplot(runif(24)))row_ha <- rowAnnotation( bar2 = expr$chr)Heatmap( mat, show_row_names = F , #cluster_rows = F , top_annotation = ha , bottom_annotation = column_ha, #对应的注释 right_annotation = row_ha)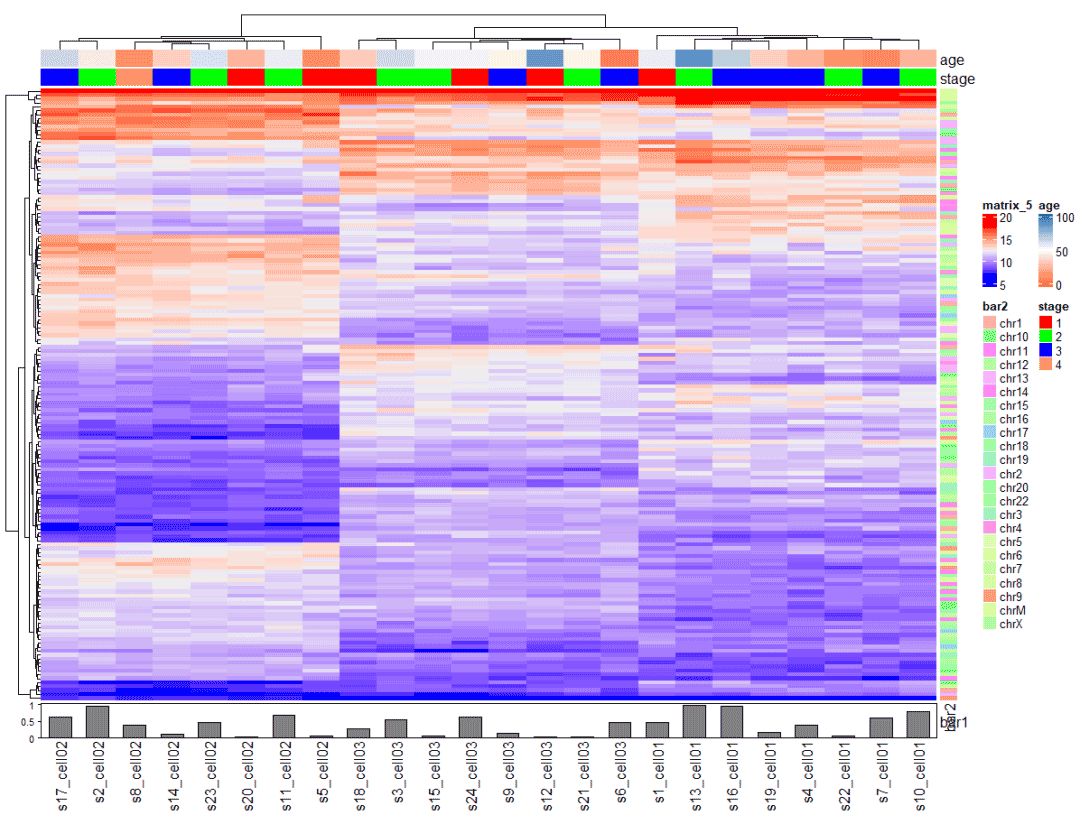
其他常用调整的函数
#cluster_rows/columns :是否进行聚类
#show_column/row_dend :是否显示聚类树
#column/row_dend_side :聚类图绘制的位置
#column_dend_height/row_dend_widht :聚类树的高度 和 宽度
三 添加“块”注释
常见的是根据聚类(kmeans等)或者 先验知识 分为几个簇,然后对簇进行注释。
3.1 k-means指定K个数
1)样本设置分为4组,基因分为3组,同时设置每个“簇”的颜色和标签
set.seed(1234)Heatmap(mat, top_annotation = HeatmapAnnotation(foo = anno_block(gp = gpar(fill = 1:4), labels = c("group1", "group2", "group3", "group4"), labels_gp = gpar(col = "white", fontsize = 10))), column_km = 4, # 列分为4个k left_annotation = rowAnnotation(foo = anno_block(gp = gpar(fill = 2:4), labels = c("group1", "group2", "group3"), labels_gp = gpar(col = "white", fontsize = 10))), row_km = 3, # show_row_names = F )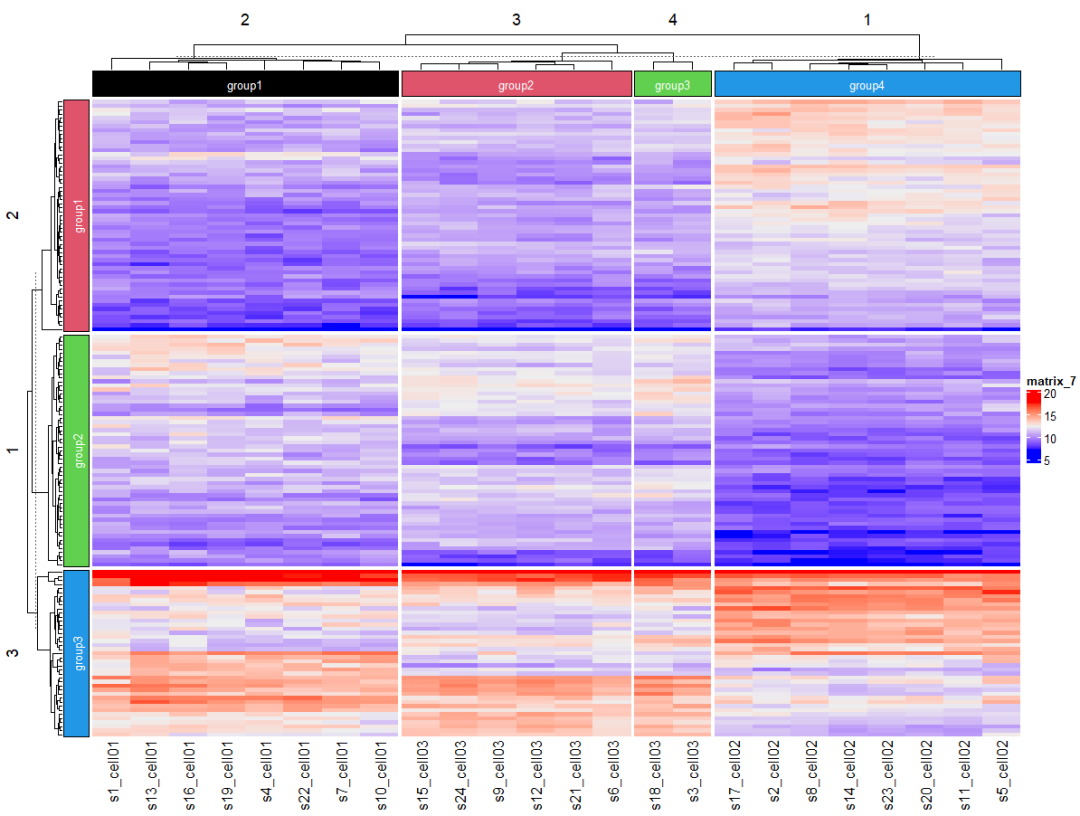
2)设置 text的颜色
Heatmap(mat, top_annotation = HeatmapAnnotation(foo = anno_block(gp = gpar(fill = 1:4), labels = c("group1", "group2", "group3", "group4"), labels_gp = gpar(col = "white", fontsize = 10))), column_km = 4, left_annotation = rowAnnotation(foo = anno_block(gp = gpar(fill = 2:4), labels = c("group1", "group2", "group3"), labels_gp = gpar(col = "white", fontsize = 10))), row_km = 3, show_row_names = F , row_title_gp = gpar( col = rainbow(5)[2:4], font = 1:3 ), row_names_gp = gpar( col = rainbow(5)[2:4], fontsize = 10:12 ), column_title_gp = gpar( fill = rainbow(5)[1:4], alpha = 0.5 ), column_names_gp = gpar( col = rainbow(5)[1:4] ) )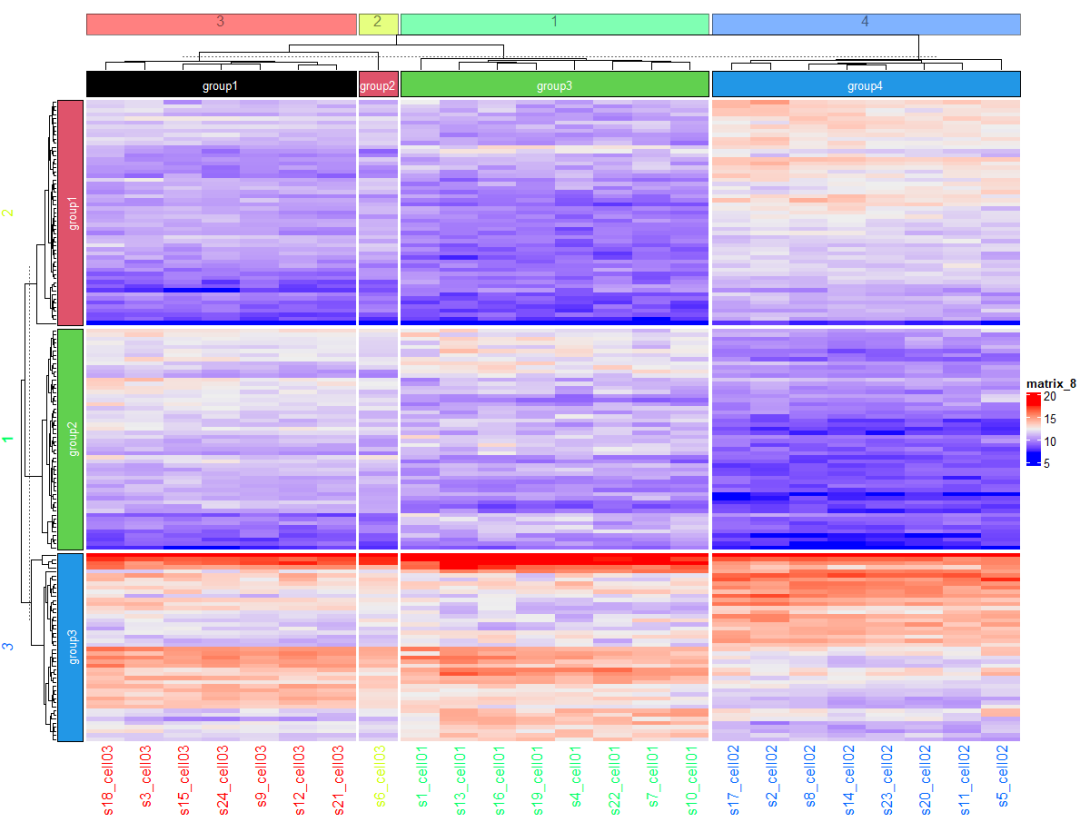
关于颜色可选#rainbow,heat.colors,terrain.colors,topo.colors,cm.colors
3.2 先验知识知道样本分为几个簇
指定样本添加列注释,假设mat中的24个样本,已知是分别为10个,10个 和4个的三组 。
实际应用中可以根据 年龄段,性别,临床分析,预后评分等指标进行的分组。
split = c( rep(c("A","B"),10) , rep("C",4) ) ha = HeatmapAnnotation(foo = anno_block(gp = gpar(fill = 2:6), labels = c("AA","BB","CC") ))col_fun = colorRamp2(c(0, 5, 10, 20), c("white", "cornflowerblue", "yellow", "red"))使用column_split 函数即可按照指定拆分
Heatmap(mat, name = "mat_cluster", column_split = split, top_annotation = ha, cluster_rows = T, cluster_columns = F, #rect_gp = gpar(col="white"), #添加白色格子线 column_title = NULL)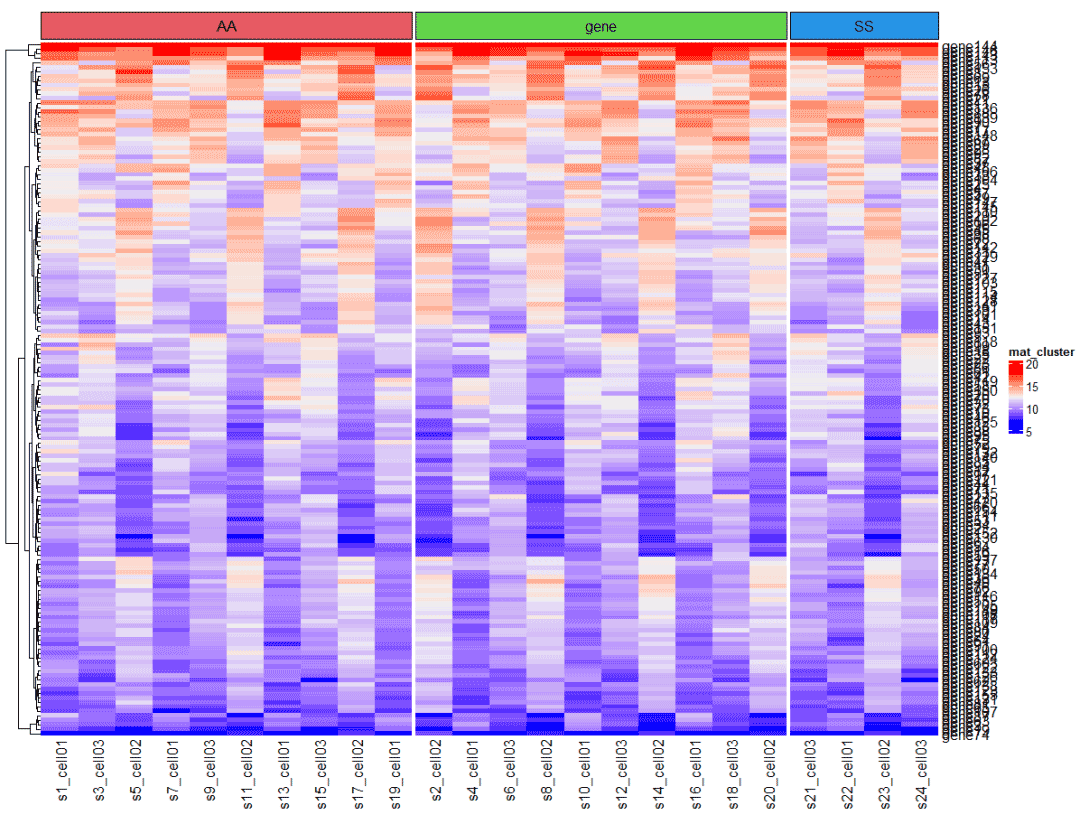
3.3 根据富集结果添加行注释
文献中经常见到 一些基因富集的通路作为 行注释的图,怎么实现呢?
1)自定义通路结果(也可以是其他想展示的内容)
group <- list( A = "Cell cycle", B = "Mismatch repair", C = "DNA replication")2)添加空白注释
ha = rowAnnotation( foo = anno_empty( border = FALSE, # 计算空白注释的宽度 width = max_text_width(unlist(group)) + unit(4, "mm")))3)通过向量拆分对应的行和列
Heatmap(mat, name = "mat", #cluster_rows = T, show_row_names = F, right_annotation = ha, row_split = c( rep(c("A","B"),30) , rep("C",95) ) , column_split = rep(c("C", "D"), 12))4)添加注释块 以及 注释文本
for(i in 1:3) { decorate_annotation( "foo", # 选择热图块 slice = i, { # 添加颜色框 grid.rect( x = 0, width = unit(2, "mm"), gp = gpar( fill = rainbow(3)[i], col = NA ), just = "left" ) # 绘制文本 grid.text( group[[i]], x = unit(4, "mm"), gp = gpar( col = rainbow(3)[i] ), just = "left") })}
需要注意的是 这里需要对应好,各位有更好的方法希望不吝告知。
四 目标基因分析
4.1 标签展示目标基因
使用anno_mark() 函数展示目标基因,至少需要两个参数,通过at 提供原始数据矩阵的索引,labels 为相应的文本标记。
1)读取待展示的基因名称,也可以是geneList的向量
name <- read.table('name.txt', header = T, check.names = FALSE)head(name)# gene#1 gene3#2 gene53#3 gene6#4 gene78#5 gene7#6 gene92)获取目标基因对应的矩阵位置;
genelist <- name$geneindex <- which(rownames(mat) %in% genelist)#得到对应的文本标签;labs <- rownames(mat)[index]3)使用labels_gp调整字体大小;
lab2 = rowAnnotation(foo = anno_mark(at = index, labels = labs, labels_gp = gpar(fontsize = 8), lines_gp = gpar()))标签展示目标基因
Heatmap(mat, name = "mat", cluster_rows = T, right_annotation = lab2, row_names_side = "right", show_row_names = F, row_names_gp = gpar(fontsize = 4))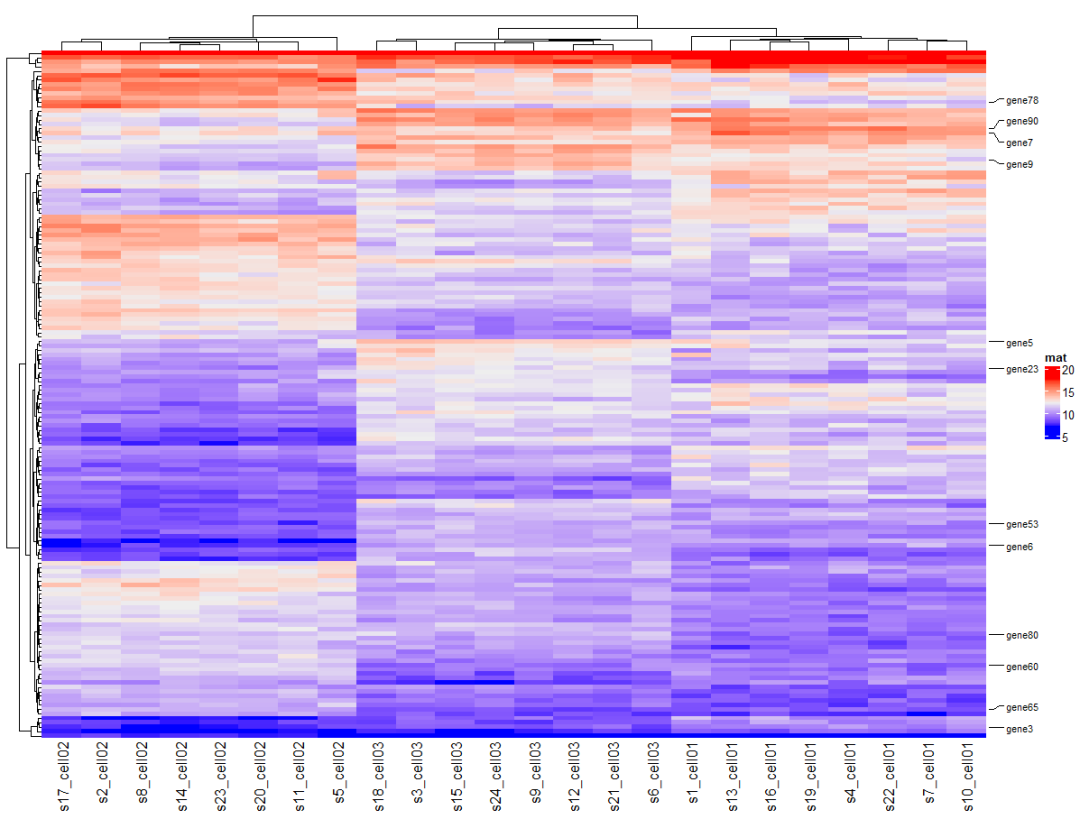
4.2 绘制目标基因热图
大部分热图存在基因太多的情况,重点展示目的基因 。
heatmap4 <- Heatmap( mat, name = "expression")heatmap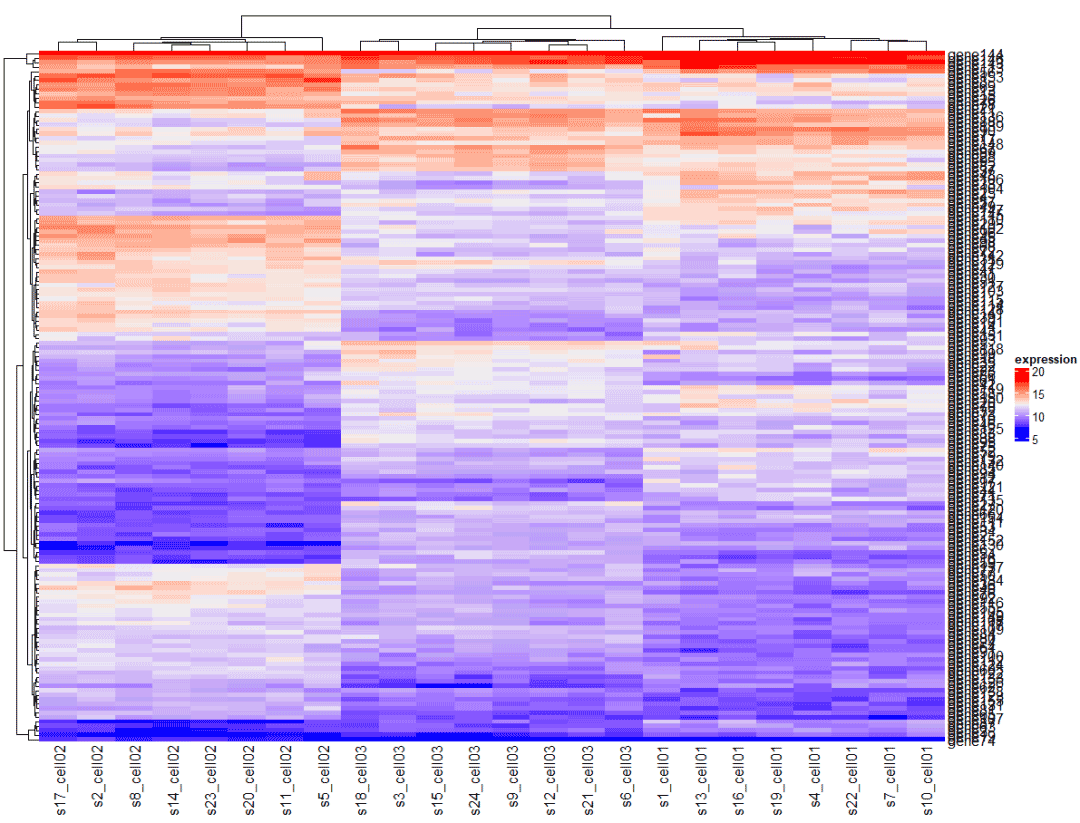
2.1 在总图中提取出来目标基因的热图,颜色与大图一致
提取目的基因所在的位置进行绘制
heatmaph5[c(1,5,6,8,9,80,144,74),]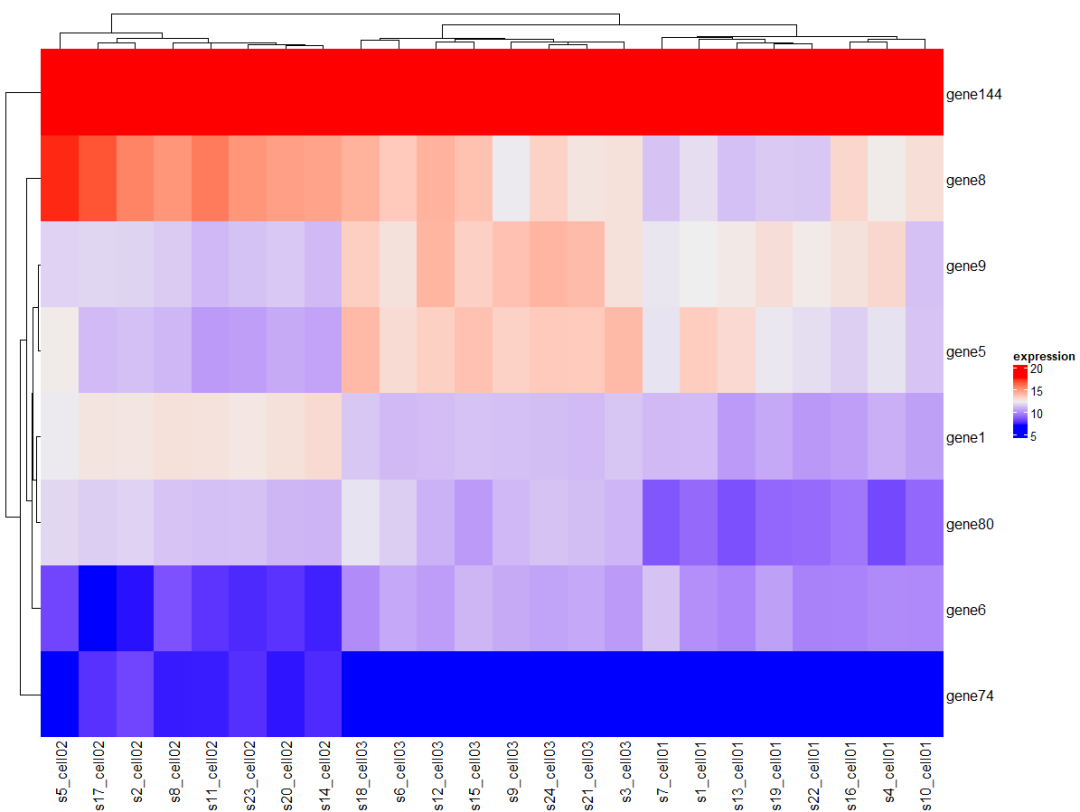
这种方式是在总的热图中直接提取目的基因的部分,热图的颜色与总的热图一致。
2.2 提取基因数据重新绘制热图
labs2 <- c("gene1", "gene5", "gene6", "gene8", "gene9", "gene80" ,"gene144", "gene74")mat2 <- as.data.frame(mat) %>% rownames_to_column("gene") %>% filter( gene %in% labs2 ) %>% column_to_rownames("gene")Heatmap(mat2)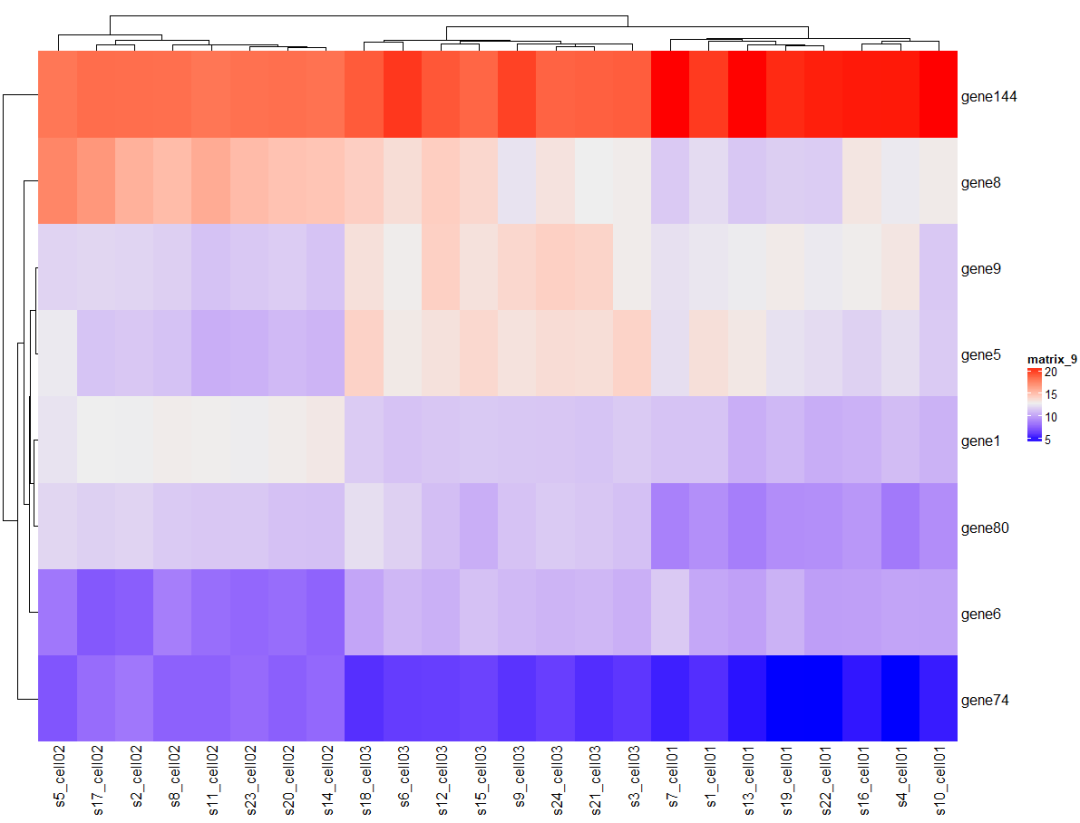
以上就是关于“怎么使用R语言ComplexHeatmap绘制复杂热图heatmap”这篇文章的内容,相信大家都有了一定的了解,希望小编分享的内容对大家有帮助,若想了解更多相关的知识内容,请关注编程网行业资讯频道。




